Python jest uniwersalnym, dynamicznym, silnie typowanym językiem z wieloma bibliotekami innych firm do zastosowań związanych z nauką danych. Obecnie są w powszechnym użyciu dwie wersje: 2 i 3. Python 2 jest „starą” wersją. Żadne nowe wersje nie są wydawane poza wersją 2.7, zapisz poprawki błędów. Python 3 to „nowa” wersja z aktywnym rozwojem. Składnia języka Python jest stosunkowo łatwa do zrozumienia w porównaniu do innych języków. Na przykład:
numbers = [1, 2, 5, 8, 9]
for number in numbers:
print(“Hello world #”, numbe
Python ma czysty wygląd dzięki regulacyjnemu podejściu do białych znaków. Choć pozornie restrykcyjny, pozwala wszystkim kodom Pythona wyglądać podobnie, co sprawia, że sprawdzanie kodu jest znacznie bardziej przewidywalne. Wszystkie pętle i warunki warunkowe (for, while, if itp.) Muszą być wcięte dla następnego bloku kodu. Popularne pakiety nauki i danych obejmują:
Numpy – szybka biblioteka tablic N-wymiarowych; podstawa wszystkich naukowych Python.
Scipy – Analiza numeryczna oparta na Numpy. Pozwala na optymalizację, algebrę liniową, transformaty Fouriera i wiele innych.
Pandas (PANel DAta) – Szybki i niezwykle elastyczny pakiet, który jest bardzo przydatny do eksploracji danych. Obsługuje dane NaN oraz szybkie indeksowanie. Obsługuje szeroką gamę zewnętrznych typów danych i formatów plików.
PYTANIA:
Co wziąć pod uwagę przed nauczeniem się nowego języka do analizy danych
Obecnie jestem na bardzo wczesnym etapie przygotowywania nowego projektu badawczego (wciąż na etapie wniosku o finansowanie) i oczekuję, że narzędzia do analizy danych, a zwłaszcza wizualizacji, odegrają rolę w tym projekcie. W związku z tym stoję przed następującym dylematem: czy powinienem nauczyć się języka Python, aby móc korzystać z jego obszernych bibliotek naukowych (Pandas, Numpy, Scipy,…), czy powinienem po prostu zanurzyć się w podobnych pakietach języka, który już znam (Rakieta, czy w mniejszym stopniu Scala)? (Idealnie uczyłbym się Pythona równolegle z korzystaniem z bibliotek statystycznych w Racket, ale nie jestem pewien, czy będę miał czas na jedno i drugie). Nie szukam odpowiedzi na ten dylemat, ale raczej opinii na temat moich różnych rozważań:
Moja obecna pozycja jest następująca:
Na korzyść Pythona:
– Intensywnie używane biblioteki
– Szeroko stosowany (może być decydujący w przypadku współpracy z innymi)
-Dużo materiałów online, aby rozpocząć naukę
-Konferencje, które są specjalnie poświęcone obliczeniom naukowym w języku Python
– Nauka Python i tak nie będzie stratą czasu
Na korzyść języka, który już znam:
-To sposób na pogłębienie mojej znajomości jednego języka zamiast powierzchownej znajomości jeszcze jednego języka (pod dewizą: powinieneś przynajmniej dobrze znać jeden język)
-To jest wykonalne. Zarówno rakieta, jak i Scala mają dobre biblioteki matematyczne i statystyczne
– Mogę od razu zacząć uczyć się tego, co muszę wiedzieć, niż najpierw nauczyć się podstaw
Dwa konkretne pytania:
- O czym zapominam?
- Na ile uciążliwy może być problem Python 2 vs 3?
ODPOWIEDZI:
Osobiście zamierzam tutaj mocno argumentować na korzyść Pythona. Jest wiele przyczyn tego, ale zamierzam oprzeć się na niektórych punktach, o których wspominali tutaj inni ludzie:
- Wybór jednego języka: zdecydowanie można mieszać i dopasowywać języki, wybierając d3 dla potrzeb wizualizacji, FORTRAN dla szybkich mnożeń macierzy i python dla całej sieci i skryptów. Możesz to zrobić wzdłuż linii, ale utrzymanie możliwie najprostszego stosu to dobry ruch, szczególnie na początku.
- Wybieranie czegoś większego od ciebie: nigdy nie chcesz naciskać na bariery języka, którego chcesz używać. Jest to ogromny problem, jeśli chodzi o języki takie jak Julia i FORTRAN, które po prostu nie oferują pełnej funkcjonalności języków takich jak Python lub R.
- Wybierz społeczność: Jedną z najtrudniejszych rzeczy do znalezienia w dowolnym języku jest społeczność. Python jest tutaj wyraźnym zwycięzcą. Jeśli utkniesz, poprosisz o coś na SO, a ktoś odpowie w ciągu kilku minut, czego po prostu nie ma w przypadku większości innych języków. Jeśli uczysz się czegoś w próżni, po prostu uczysz się dużo wolniej.
Jeśli chodzi o punkty ujemne, mogę je odepchnąć. Pogłębienie znajomości jednego języka jest dobrym pomysłem, ale znajomość tylko jednego języka bez praktyki uogólniania tej wiedzy na inne języki jest dobrym sposobem na zastrzelenie się w stopę. Zmieniłem cały mój ulubiony stos programistyczny trzy razy przez wiele lat, przechodząc z MATLAB na Javę na Haskell na Python. Nauka przenoszenia wiedzy na inny język jest o wiele bardziej cenna niż znajomość jednego. O ile to wykonalne, będzie to coś, co zobaczysz wielokrotnie w każdej karierze programistycznej. Kompletność Turinga oznacza, że możesz technicznie zrobić wszystko z HTML4 i CSS3, ale chcesz wybrać odpowiednie narzędzie do pracy. Jeśli zobaczysz idealne narzędzie i zdecydujesz się je zostawić na poboczu, zwolnisz, żałując, że nie masz narzędzi, które zostawiłeś. Świetnym przykładem tego ostatniego punktu jest próba wdrożenia kodu R. Ogromnie brakuje możliwości sieciowych R w porównaniu do Pythona, a jeśli chcesz wdrożyć usługę lub użyć nieco nietypowych pakietów ścieżek, fakt, że pip ma o rząd wielkości więcej pakietów niż CRAN, jest ogromną pomocą .
Z mojego doświadczenia wynika, że o platformie analizy danych należy pamiętać:
- Czy może obsłużyć rozmiar potrzebnych mi danych? Jeśli twoje zestawy danych mieszczą się w pamięci, zwykle nie ma większych problemów, chociaż AFAIK Python jest nieco bardziej wydajny pod względem pamięci niż R. Jeśli potrzebujesz obsługiwać zestawy danych większe niż pamięć, platforma musi sobie z tym poradzić. W takim przypadku SQL obejmuje podstawowe statystyki, Python + Apache Spark to kolejna opcja.
- Czy platforma pokrywa wszystkie moje potrzeby w zakresie analizy? Największą irytacją, jaką spotkałem w projektach eksploracji danych, jest konieczność żonglowania kilkoma narzędziami, ponieważ narzędzie A dobrze radzi sobie z połączeniami internetowymi, narzędzie B wykonuje statystyki, a narzędzie C wyświetla ładne zdjęcia. Chcesz, aby wybrana przez ciebie broń obejmowała jak najwięcej aspektów twoich projektów. Rozważając ten problem, Python jest bardzo obszerny, ale R ma wiele wbudowanych testów statystycznych gotowych do użycia, jeśli tego potrzebujesz.
Narzędzia i protokół do powtarzalnej analizy danych za pomocą Pythona.
Pracuję nad projektem nauki danych przy użyciu Pythona. Projekt składa się z kilku etapów. Każdy etap obejmuje pobranie zestawu danych, użycie skryptów Python, danych pomocniczych, konfiguracji i parametrów oraz utworzenie innego zestawu danych. Przechowuję kod w git, więc ta część jest objęta. Chciałbym usłyszeć o:
- Narzędzia do kontroli wersji danych.
- Narzędzia umożliwiające odtworzenie etapów i eksperymentów.
- Protokół i sugerowana struktura katalogów dla takiego projektu.
- Zautomatyzowane narzędzia do budowania / uruchamiania.
Odkąd zacząłem prowadzić badania w środowisku akademickim, ciągle szukałem satysfakcjonującego przepływu pracy. Myślę, że w końcu znalazłem coś, z czego jestem zadowolony:
1) Poddaj wszystko kontroli wersji, np. Git: W projektach badań hobby korzystam z GitHub, do badań w pracy korzystam z prywatnego GitLab serwer dostarczany przez naszą uczelnię. Tam też przechowuję moje zbiory danych.
2) Większość moich analiz wykonuję wraz z dokumentacją dotyczącą notebooków IPython. Dla mnie bardzo dobrze jest zorganizować kod, wykresy i dyskusję / wnioski w jednym dokumencie. Jeśli uruchamiam większe skrypty, zwykle umieszczam je w osobnych plikach skryptowych .py, ale nadal je wykonuję z notesu IPython poprzez magię% run, aby dodać informacje o celu, wyniku i innych parametrach. Napisałem małe rozszerzenie o komórkowej magii dla notebooków IPython i IPython, zwane „znakiem wodnym”, którego używam do wygodnego tworzenia znaczników czasu i śledzenia
Najlepszym narzędziem odtwarzalności jest rejestrowanie twoich działań, mniej więcej tak: eksperyment / wejście; spodziewany ; obserwacja / wynik; aktualna hipoteza i jeśli jest obsługiwana lub odrzucona exp1; oczekiwany 1; obs1; obsługiwana kilka fantazyjnych hipotez Można to zapisać na papierze, ale jeśli twoje eksperymenty mieszczą się w środowisku obliczeniowym, możesz użyć narzędzi obliczeniowych do częściowej lub całkowitej automatyzacji tego procesu rejestrowania (szczególnie poprzez pomoc w śledzeniu wejściowych zestawów danych, które mogą być ogromne, a dane wyjściowe). Świetnym narzędziem odtwarzalności dla Pythona o niskiej krzywej uczenia się jest oczywiście Notatnik IPython / Jupyter (nie zapomnij o% logon i% logstart magics). Innym doskonałym narzędziem, które jest bardzo aktualne (2015), jest przepis, który jest bardzo podobny do sumatry (patrz poniżej), ale został stworzony specjalnie dla Pythona. Nie wiem, czy to działa z notatnikami Jupyter, ale wiem, że autor często z nich korzysta, więc sądzę, że jeśli nie jest obecnie obsługiwany, będzie w przyszłości. Git jest także niesamowity i nie jest powiązany z Pythonem. Pomoże ci to nie tylko zachować historię wszystkich twoich eksperymentów, kodu, zestawów danych, rycin itp., Ale także zapewni ci narzędzia do utrzymywania (git kilof), współpracy (obwinianie) i debugowania (git-bisect) przy użyciu naukowych metoda debugowania (zwana debugowaniem delta). Oto historia fikcji
badacz próbuje stworzyć własny system rejestrowania eksperymentów, dopóki nie stanie się on kopią Gita. Innym ogólnym narzędziem współpracującym z dowolnym językiem (z API Pythona na pypi) jest Sumatra, która została specjalnie zaprojektowana, aby pomóc Ci w przeprowadzaniu badań powtarzalnych (powtarzalnych)
Ma na celu uzyskanie takich samych wyników, biorąc pod uwagę dokładnie ten sam kod i oprogramowanie, podczas gdy odtwarzalność ma na celu uzyskanie takich samych wyników dla dowolnego nośnika, co jest o wiele trudniejsze, czasochłonne i niemożliwe do zautomatyzowania. Oto jak działa Sumatra: dla każdego eksperymentu przeprowadzanego przez Sumatrę oprogramowanie to będzie działać jak „zapisywanie stanu gry” często występujące w grach wideo. Dokładniej, pozwoli to zaoszczędzić:
* wszystkie parametry, które podałeś;
* dokładny stan kodu źródłowego całej eksperymentalnej aplikacji i plików konfiguracyjnych;
* dane wyjściowe / wykresy / wyniki, a także każdy plik wygenerowany przez aplikację eksperymentalną.
Następnie zbuduje bazę danych ze znacznikiem czasu i innymi metadatami dla każdego eksperymentu, które można później przeszukiwać za pomocą webGUI. Ponieważ Sumatra zapisała pełny stan aplikacji dla określonego eksperymentu w jednym określonym momencie, możesz przywrócić kod, który dał określony wynik w dowolnym momencie, dzięki czemu masz powtarzalne badania przy niskim koszcie (z wyjątkiem przechowywania, jeśli pracujesz na ogromnych zestawach danych, ale możesz skonfigurować wyjątki, jeśli nie chcesz zapisywać wszystkiego za każdym razem).
W końcu możesz użyć Git lub Sumatry, zapewnią one tę samą moc powtarzalności, ale Sumatra jest specjalnie dostosowana do badań naukowych, więc zapewnia kilka fantazyjnych narzędzi, takich jak internetowy interfejs GUI, do indeksowania twoich wyników, podczas gdy Git jest bardziej dostosowany do obsługi kodu (ale ma narzędzia do debugowania, takie jak git-bisect, więc jeśli masz eksperymenty obejmujące kody, może być lepiej). Lub oczywiście możesz użyć obu! / EDIT: dsign dotknął tutaj bardzo ważnej kwestii: powtarzalność konfiguracji jest równie ważna jak replikacja aplikacji. Innymi słowy, powinieneś przynajmniej podać pełną listę używanych bibliotek i kompilatorów wraz z ich dokładnymi wersjami i szczegóły twojej platformy. Osobiście w informatyce naukowej w Pythonie odkryłem, że pakowanie aplikacji wraz z bibliotekami jest po prostu zbyt bolesne, dlatego teraz po prostu używam uniwersalnego naukowego pakietu python, takiego jak Anaconda (z doskonałym menedżerem pakietów conda), i po prostu doradzam użytkownikom korzystanie z tego samego pakietu. Innym rozwiązaniem może być dostarczenie skryptu do automatycznego generowania virtualenv lub spakowanie wszystkiego przy użyciu komercyjnej aplikacji Docker, cytowanej przez dsign lub Vagrant z open source (na przykład pylearn2-in-a-box, które używają Vagrant do stworzenia łatwo redystrybucyjnej dystrybucji pakiet środowiska wirtualnego). / EDIT2: Oto świetne wideo podsumowujące (do debugowania, ale można je również zastosować do badań), co jest fundamentem do przeprowadzenia powtarzalnych badań: rejestrowanie eksperymentów i poszczególnych etapów metody naukowej, rodzaj „jawnego eksperymentowania”.
Koniecznie sprawdź dokera! Ogólnie rzecz biorąc, wszystkie inne dobre rzeczy, które inżynieria oprogramowania tworzyła przez dziesięciolecia w celu zapewnienia izolacji i odtwarzalności. Chciałbym podkreślić, że nie wystarczy mieć powtarzalne przepływy pracy, ale także łatwe do odtworzenia przepływy pracy. Pokażę, co mam na myśli. Załóżmy, że twój projekt używa Pythona, bazy danych X i Scipy. Z pewnością będziesz używać konkretnej biblioteki do łączenia się z bazą danych z Pythona, a Scipy z kolei będzie używać rzadkich procedur algebraicznych. Jest to z pewnością bardzo prosta konfiguracja, ale nie do końca łatwa w konfiguracji, przewidziana gra słów. Jeśli ktoś chce wykonać twoje skrypty, będzie musiał zainstalować wszystkie zależności. Lub, co gorsza, mogła mieć już zainstalowane niekompatybilne wersje. Naprawienie tych rzeczy wymaga czasu. Zajmie ci to również trochę czasu, jeśli będziesz musiał przenieść swoje obliczenia do klastra, innego klastra lub niektórych serwerów w chmurze. Tutaj uznaję dokera za użyteczny. Docker to sposób na sformalizowanie i skompilowanie przepisów dla środowisk binarnych. Możesz zapisać następujące pliki w pliku dockerfile (używam tutaj zwykłego angielskiego zamiast składni Dockerfile):
* Zacznij od podstawowego środowiska binarnego, takiego jak Ubuntu
* Zainstaluj libsparse-dev
* Zainstaluj numpy i scipy
* Zainstaluj X
* Zainstaluj libX-dev
* Zainstaluj Python-X
* Zainstaluj IPython-Notebook
* Skopiuj moje skrypty / notesy Pythona do mojego środowiska binarnego, tych plików danych i tych konfiguracji, aby wykonywać inne czynności. Aby zapewnić odtwarzalność, skopiuj je z nazwanego adresu URL zamiast pliku lokalnego.
* Może uruchomić IPython-Notebook.
Niektóre linie będą instalowały różne rzeczy w Pythonie przy użyciu pip, ponieważ pip może wykonać bardzo czystą pracę przy wyborze konkretnych wersji pakietów. Sprawdź to też! I to wszystko. Jeśli po utworzeniu pliku Dockerfile można go zbudować, może go zbudować w dowolnym miejscu, przez kogokolwiek (pod warunkiem, że mają one również dostęp do plików specyficznych dla projektu, np. Ponieważ umieścisz je w publicznym adresie URL, do którego odwołuje się plik Dockerfile). Co jest najlepsze, może przesłać powstałe środowisko (zwane „obrazem”) na publiczny lub prywatny serwer (zwany „rejestrem”), z którego mogą korzystać inne osoby. Tak więc, kiedy publikujesz swój przepływ pracy, masz zarówno w pełni powtarzalny przepis w postaci pliku Docker, jak i łatwy sposób dla ciebie lub innych osób na odtworzenie tego, co robisz:
docker run dockerregistery.thewheezylab.org/nowyouwillbelieveme
Lub jeśli chcą się bawić w twoje skrypty i tak dalej:
dockerregistery.thewheezylab.org/nowyouwillbelieveme /bin/bash
Gradient stochastyczny w oparciu o operacje wektorowe?
Załóżmy, że chcę trenować algorytm regresji spadku gradientu stochastycznego przy użyciu zestawu danych zawierającego N próbek. Ponieważ rozmiar zestawu danych jest ustalony, ponownie użyję danych T razy. Przy każdej iteracji lub „epoce” używam każdej próbki treningowej dokładnie raz po losowym uporządkowaniu całego zestawu treningowego. Moja implementacja oparta jest na Pythonie i Numpy. Dlatego przy użyciu operacji wektorowych może znacznie skrócić czas obliczeń. Wymyślenie wektorowej implementacji opadania gradientu wsadowego jest dość proste. Jednak w przypadku stochastycznego spadku gradientu nie mogę dowiedzieć się, jak uniknąć zewnętrznej pętli, która przechodzi przez wszystkie próbki w każdej epoce. Czy ktoś zna wektoryzowaną implementację stochastycznego spadku gradientu?
EDYCJA: Zostałem zapytany, dlaczego chciałbym korzystać z opadania gradientu online, jeśli rozmiar mojego zestawu danych jest stały.
Opadanie gradientu online zbiega się wolniej niż opadanie gradientu wsadowego do minimum kosztów empirycznych. Jednak zbiega się szybciej do minimum oczekiwanego kosztu, który mierzy wydajność uogólnienia. Chciałbym przetestować wpływ tych teoretycznych wyników w moim konkretnym problemie za pomocą weryfikacji krzyżowej. Bez implementacji wektoryzacyjnej mój kod zejścia gradientowego w trybie online jest znacznie wolniejszy niż kod partii z gradientowym spadkiem. To znacznie wydłuża czas potrzebny na zakończenie procesu weryfikacji krzyżowej.
Method: on-line gradient descent (regression)
Input: X (nxp matrix; each line contains a training sample, represented as a length-p vector), Y (length-n vector; Output: A (length-p+1 vector of coefficients)
Initialize coefficients (assign value 0 to all coefficients)
Calculate outputs F
prev_error = inf
error = sum((F-Y)^2)/n
it = 0
while abs(error – prev_error)>ERROR_THRESHOLD and it<=MAX_ITERATIONS:
Randomly shuffle training samples
for each training sample i:
Compute error for training sample i
Update coefficients based on the error above
prev_error = error
Calculate outputs F
error = sum((F-Y)^2)/n
it = it + 1
Po pierwsze, słowo „próbka” jest zwykle używane do opisania podgrupy populacji, więc będę odnosił się do tego samego co „przykład”. Twoja implementacja SGD jest powolna z powodu tej linii:
for each training example i:
Tutaj jawnie używasz dokładnie jednego przykładu dla każdej aktualizacji parametrów modelu. Z definicji wektoryzacja jest techniką przekształcania operacji na jednym elemencie na operacje na wektorze takich elementów. Dlatego nie możesz przetwarzać przykładów jeden po drugim i nadal używać wektoryzacji. Możesz jednak oszacować rzeczywistą wartość SGD za pomocą mini-partii. Mini-partia to niewielki podzbiór oryginalnego zestawu danych (powiedzmy 100 przykładów). Obliczasz błędy i aktualizacje parametrów na podstawie mini-partii, ale wciąż iterujesz wiele z nich bez globalnej optymalizacji, co czyni proces stochastycznym. Aby więc znacznie przyspieszyć wdrożenie, wystarczy zmienić poprzednią linię na:
batches = split dataset into mini-batches
for batch in batches:
i obliczyć błąd z partii, a nie z jednego przykładu. Choć dość oczywiste, powinienem również wspomnieć o wektoryzacji na poziomie poszczególnych przykładów. Oznacza to, że zamiast czegoś takiego:
theta = np.array([…]) # parameter vector
x = np.array([…]) # example
y = 0 # predicted response
for i in range(len(example)):
y += x[i] * theta[i]
error = (true_y – y) ** 2 # true_y – true value of response
you should definitely do something like this:
error = (true_y – sum(np.dot(x, theta))) ** 2
which, again, easy to generalize for mini-batches:
true_y = np.array([…]) # vector of response values
X = np.array([[…], […]]) # mini-batch
errors = true_y – sum(np.dot(X, theta), 1)
error = sum(e ** 2 for e in errors)
Sprawdź metodę częściowego dopasowania klasyfikatora SGD firmy scikit. Masz kontrolę nad tym, co nazywasz: możesz to zrobić „prawdziwą” nauką online, przekazując instancję na raz, lub możesz grupować instancje w mini-partie, jeśli wszystkie dane są dostępne w tablicy. Jeśli tak, możesz pokroić tablicę, aby uzyskać minibatche.
Problem optymalizacji szeregów czasowych Pandy: dodaj rok
Mam pand DataFrame zawierającą kolumnę szeregów czasowych. Lata są przesunięte w przeszłości, więc muszę dodać stałą liczbę lat do każdego elementu tej kolumny. Najlepszym sposobem, jaki znalazłem, jest iteracja wszystkich zapisów i wykorzystanie
x.replace (year = x.year + years) # x = bieżący element, lata = lata do dodania Jest cytonizowany jak poniżej, ale nadal bardzo wolny (proofing)
cdef list _addYearsToTimestamps(list elts, int years):
cdef cpdatetime x
cdef int i
for (i, x) in enumerate(elts):
try:
elts[i] = x.replace(year=x.year + years)
except Exception as e:
logError(None, “Cannot replace year of %s – leaving value as this: %s” % (str(x), repr(e)))
return elts
def fixYear(data):
data.loc[:, ‘timestamp’] = _addYearsToTimestamps(list(data.loc[:, ‘timestamp’]), REAL_YEAR-(list(data[-1:][‘return data
Jestem pewien, że istnieje sposób na zmianę roku bez iteracji, korzystając z funkcji Pandas Timestamp. Niestety nie wiem jak. Czy ktoś mógłby opracować?
Utwórz obiekt Pimed Timedelta, a następnie dodaj za pomocą operatora + =:
x = pandas.Timedelta(days=365)
mydataframe.timestampcolumn += x
Kluczem jest więc przechowywanie szeregów czasowych jako znaczników czasu. Aby to zrobić, użyj funkcji pandy to_datetime:
mydataframe[‘timestampcolumn’] = pandas.to_datetime(x[‘epoch’], unit=’s’)
zakładając, że masz znaczniki czasu jako epokowe sekundy w ramce danych x. Oczywiście nie jest to wymóg; zobacz dokumentację to_datetime, aby przekonwertować inne formaty.
Oto implementacja rozwiązania i demonstracja.
#!/usr/bin/env python3
import random
import pandas
import time
import datetime
def getRandomDates(n):
tsMin = time.mktime(time.strptime(“1980-01-01 00:00:00”, “%Y-%m-%d %H:%M:%S”))
tsMax = time.mktime(time.strptime(“2005-12-31 23:59:59”, “%Y-%m-%d %H:%M:%S”))
return pandas.Series([datetime.datetime.fromtimestamp(tsMin + random.random() * (tsMax – tsMin)) for x in def setMaxYear(tss, target):
maxYearBefore = tss.max().to_datetime().year
# timedelta cannot be given in years, so we compute the number of days to add in the next line
deltaDays = (datetime.date(target, 1, 1) – datetime.date(maxYearBefore, 1, 1)).days
return tss + pandas.Timedelta(days=deltaDays)
data = pandas.DataFrame({‘t1’: getRandomDates(1000)})
data[‘t2’] = setMaxYear(data[‘t1’], 2015)
data[‘delta’] = data[‘t2’] – data[‘t1’]
print(data)
print(“delta min: %s” % str(min(data[‘delta’])))
print(“delta max: %s” % str(max(data[‘delta’])))
Jak rozpoznać dwuczęściowy termin, gdy przestrzeń zostanie usunięta?
(„Bigdata” i „big data”)
Nie jestem facetem NLP i mam to pytanie.
Mam tekstowy zestaw danych zawierający terminy, które wyglądają jak „duże dane” i „duże dane”. Dla mnie oba są takie same. Jak mogę je wykryć w języku NLTK (Python)? Lub jakikolwiek inny moduł NLP w Pythonie?
Jest miła implementacja tego w gensim:
http://radimrehurek.com/gensim/models/phrases.html
Zasadniczo wykorzystuje podejście oparte na danych do wykrywania fraz, tj. wspólne kolokacje. Jeśli więc nakarmisz klasę fraz kilka zdań, a wyrażenie „duże dane” pojawi się bardzo często, klasa nauczy się łączyć „big data” w pojedynczy token „big_data”.
Wyodrębnianie obrazów w Pythonie
W mojej klasie muszę utworzyć aplikację przy użyciu dwóch klasyfikatorów, aby zdecydować, czy obiekt na obrazie jest przykładem phylum porifera (seasponge) czy jakiegoś innego obiektu. Jestem jednak całkowicie zagubiony, jeśli chodzi o techniki ekstrakcji funkcji w pythonie. Mój doradca przekonał mnie do korzystania ze zdjęć, które nie były omówione w klasie. Czy ktoś może skierować mnie w stronę sensownej dokumentacji lub lektury lub zasugerować metody do rozważenia?
Ten świetny samouczek obejmuje podstawy neuronowych układów splotowych, które obecnie osiągają najnowszą wydajność w większości zadań związanych z widzeniem: http://deeplearning.net/tutorial/lenet.html
Istnieje wiele opcji dla CNN w Pythonie, w tym Theano i biblioteki zbudowane na nim (uważam, że keras jest łatwy w użyciu).
Jeśli wolisz unikać głębokiego uczenia się, możesz zajrzeć do OpenCV, który może nauczyć się wielu innych rodzajów funkcji, kaskad Haar i funkcji SIFT.
Parametry Hypertuning XGBoost
XGBoost wykonał świetną robotę, jeśli chodzi o radzenie sobie zarówno z kategorycznymi, jak i ciągłymi zmiennymi zależnymi. Ale jak wybrać zoptymalizowane parametry dla problemu XGBoost?
Oto jak zastosowałem parametry do ostatniego problemu Kaggle:
param <- list( objective = “reg:linear”,
booster = “gbtree”,
eta = 0.02, # 0.06, #0.01,
max_depth = 10, #changed from default of 8
subsample = 0.5, # 0.7
colsample_bytree = 0.7, # 0.7
num_parallel_tree = 5
# alpha = 0.0001,
# lambda = 1
)
clf <- xgb.train( params = param,
data = dtrain,
nrounds = 3000, #300, #280, #125, #250, # changed from 300
verbose = 0,
early.stop.round = 100,
watchlist = watchlist,
maximize = FALSE,
feval=RMPSE
)
Wszystko, co robię, aby eksperymentować, to losowo (intuicyjnie) wybrać inny zestaw parametrów, aby poprawić wynik. Czy w ogóle automatyzuję wybór zoptymalizowanego (najlepszego) zestawu parametrów? (Odpowiedzi mogą być w dowolnym języku. Po prostu szukam techniki)
Ilekroć pracuję z xgboost, często dokonuję własnego wyszukiwania parametrów homebrew, ale możesz to zrobić za pomocą pakietu caret, takiego jak wspomniany wcześniej KrisP.
- Caret
Zobacz tę odpowiedź na Cross Validated, aby uzyskać dokładne wyjaśnienie, jak korzystać z pakietu Caret do wyszukiwania hiperparametrów na xgboost. Jak dostroić hiperparametry drzew xgboost?
- Niestandardowe wyszukiwanie w siatce
Często zaczynam od kilku założeń opartych na slajdach Owena Zhanga na temat wskazówek dotyczących analizy danych
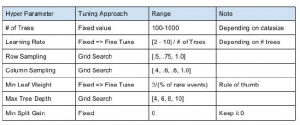
Tutaj możesz zobaczyć, że przede wszystkim musisz dostroić próbkowanie wierszy, próbkowanie kolumn i być może maksymalną głębokość drzewa. Oto jak wykonuję niestandardowe próbkowanie wierszy i próbkowanie kolumn w poszukiwaniu problemu, nad którym obecnie pracuję:
searchGridSubCol <- expand.grid (podpróbka = c (0,5, 0,75, 1),
colsample_bytree = c (0,6, 0,8, 1))
ntrees <- 100
#Buduj obiekt xgb.DMatrix
DMMatrixTrain <- xgb.DMatrix (data = yourMatrix, label = yourTarget)
rmseErrorsHyperparameters <- zastosuj (searchGridSubCol, 1, funkcja (parameterList) {
#Extract Parametry do przetestowania
currentSubsampleRate <- parameterList [[„subsample”]]
currentColsampleRate <- parameterList [[“colsample_bytree”]]
xgboostModelCV <- xgb.cv (data = DMMatrixTrain, nrounds = ntrees, nfold = 5, showsd = TRUE,
metrics = “rmse”, verbose = TRUE, “eval_metric” = “rmse”,
„cel” = „reg: liniowy”, „maks. głębokość” = 15, „eta” = 2 / ntrees,
„subsample” = currentSubsampleRate, „colsample_bytree” = currentColsampleRate)
xvalidationScores <- as.data.frame (xgboostModelCV)
# Zapisz rmse ostatniej iteracji
rmse <- tail (xvalidationScores $ test.rmse.mean, 1)
return (c (rmse, currentSubsampleRate, currentColsampleRate))
})
W połączeniu z pewną magią ggplot2, korzystając z wyników tej funkcji zastosuj, możesz wykreślić graficzną reprezentację wyszukiwania.
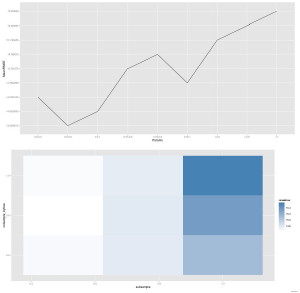
Na tym wykresie jaśniejsze kolory oznaczają mniejszy błąd, a każdy blok reprezentuje unikalną kombinację próbkowania kolumn i próbkowania wierszy. Więc jeśli chcesz przeprowadzić dodatkowe wyszukiwanie powiedzmy eta (lub głębokość drzewa), skończysz z jednym z tych wykresów dla każdego testowanego parametru eta. Widzę, że masz inną metrykę oceny (RMPSE), po prostu podłącz ją do funkcji weryfikacji krzyżowej, a otrzymasz pożądany wynik. Poza tym nie martwiłbym się zbytnio dopracowaniem innych parametrów, ponieważ nie poprawi to zbytnio wydajności, a przynajmniej nie tak dużo w porównaniu do poświęcania więcej czasu na funkcje techniczne lub czyszczenie danych.
- Inne
Możliwe jest również wyszukiwanie losowe i wybór parametrów bayesowskich, ale jeszcze nie znalazłem ich implementacji.
Budowanie modelu uczenia maszynowego w celu przewidywania plonów na podstawie danych środowiskowych
Mam zestaw danych zawierający dane dotyczące temperatury, opadów i plonów soi dla gospodarstwa przez 10 lat (2005–2014). Na podstawie tych danych chciałbym przewidzieć rentowności w 2015 r. Należy pamiętać, że zestaw danych zawiera DZIENNE wartości temperatury i opadów, ale tylko 1 wartość rocznie dla plonu, ponieważ zbiory plonów odbywają się pod koniec sezonu wegetacyjnego zbiorów. Chcę zbudować regresję lub inny model oparty na uczeniu maszynowym, aby przewidzieć plony w 2015 r., W oparciu o regresję / jakiś inny model wyprowadzony z badania zależności między wydajnościami a temperaturą i opadami w poprzednich latach.
Znam się na uczeniu maszynowym za pomocą scikit-learn. Nie wiem jednak, jak przedstawić ten problem. Problem polega na tym, że temperatura i opady są codzienne, ale wydajność wynosi tylko 1 wartość rocznie. Jak do tego podejść?
Na początek możesz przewidzieć wydajność w nadchodzącym roku na podstawie dziennych danych z poprzedniego roku. Możesz oszacować parametry modelu, biorąc pod uwagę wartość każdego roku jako jeden „punkt”, a następnie zweryfikuj model za pomocą walidacji krzyżowej. Możesz to przedłużyć model, biorąc pod uwagę więcej niż w ubiegłym roku, ale spójrz za daleko, a będziesz mieć problemy z weryfikacją modelu i stroju.
Popraw dokładność k-średnich
Nasza broń:
Eksperymentuję z K-średnich i Hadoopem, gdzie jestem przywiązany do tych opcji z różnych powodów (np. Pomóż mi wygrać tę wojnę!).
Pole bitwy:
Mam artykuły należące do kategorii c, w których c jest naprawione. Wektoryzuję zawartość artykułów do funkcji TF-IDF. Teraz używam naiwnego algorytmu k-średnich, który zaczyna c centroidy i rozpoczyna iteracyjnie grupowanie artykułów (tj. Wiersze macierzy TF-IDF, gdzie można zobaczyć, jak to zbudowałem), aż do pojawienia się converenge.
Specjalne notatki:
- Początkowe centroidy: Wypróbowane losowo z każdej kategorii lub ze średniej wszystkich artykułów z każdej kategorii.
- Funkcja odległości: euklidesowa.
Dokładność jest niska, zgodnie z oczekiwaniami, czy mogę zrobić coś lepszego, dokonując innego wyboru dla początkowych centroidów, i / lub wybierając inną funkcję odległości?
print „Hello Data Science site!” 🙂
Sposób skonfigurowania tego eksperymentu ma dla mnie sens z punktu widzenia projektowania, ale myślę, że jest jeszcze kilka aspektów, które można jeszcze zbadać. Po pierwsze, możliwe, że nieinformacyjne funkcje odwracają uwagę twojego klasyfikatora, prowadząc do gorszych wynikówi. W analizie tekstu często mówimy o zatrzymaniu filtrowania słów, która jest po prostu procesem usuwania takiego tekstu (np., I, lub, itp.). Istnieją standardowe listy słów stop, które można łatwo znaleźć w Internecie (np. Ten), ale czasami mogą być trudne. Najlepszym rozwiązaniem jest zbudowanie tabeli powiązanej z częstotliwością funkcji w zależności od klasy, ponieważ uzyska ona funkcje specyficzne dla domeny, których prawdopodobnie nie znajdziesz w takich tabelach przeglądowych. Istnieją różne dowody na skuteczność usuwania słów zatrzymanych w literaturze, ale myślę, że te odkrycia dotyczą głównie specyficznych dla klasyfikatora (na przykład, na maszynach wektorów pomocniczych zwykle mniej wpływają nieinformacyjne cechy niż naiwne Bayes klasyfikator. Podejrzewam, że k-średnie należy do tej drugiej kategorii). Po drugie, możesz rozważyć inne podejście do modelowania obiektów niż tf-idf. Nic przeciwko tf-idf – działa dobrze w przypadku wielu problemów – ale lubię zacząć od modelowania funkcji binarnych, chyba że mam eksperymentalne dowody wskazujące, że bardziej złożone podejście prowadzi do lepszych wyników. To powiedziawszy, możliwe jest, że k-średnie mogłyby dziwnie zareagować na zmianę z przestrzeni zmiennoprzecinkowej na binarną. Jest to z pewnością łatwo sprawdzalna hipoteza! Na koniec możesz spojrzeć na oczekiwany rozkład klas w swoim zestawie danych. Czy wszystkie klasy są jednakowo prawdopodobne? Jeśli nie, możesz uzyskać lepsze wyniki albo z próbkowania, albo z innej miary odległości. Wiadomo, że k-średnich reaguje słabo w przekrzywionych sytuacjach klasowych, więc jest to również kwestia do rozważenia! Prawdopodobnie w Twojej domenie dostępne są badania opisujące, jak inni poradzili sobie z tą sytuacją.